![[의료 이미지] CT image 이해하기 (feat. Dicom 파일)](https://img1.daumcdn.net/thumb/R750x0/?scode=mtistory2&fname=https%3A%2F%2Fblog.kakaocdn.net%2Fdn%2FNZX1F%2Fbtq7EAA7veE%2FjMMCsYGGihkRZOycz3uzD0%2Fimg.jpg)

논문을 읽기 앞서 CT image의 Dicom 파일과 뇌출혈 subtype에 관해 알아보자.
1. Dicom 파일이란?
DICOM은 의학 분야의 Digital Imaging and Communications의 약자이다. 초음파 및 MRI 이미지와 같은 의료 정보를 환자의 정보와 함께 하나의 Dicom 파일에 저장할 수 있다.
- dicom 파일 여는 법
pydicom libraray를 설치 후에 사용 할 수 있다. 데이터 안에는 다음과 같은 meta 정보가 포함되어 있다.
meta 정보에는 이미지 사이즈, Window 사이즈, 환자 정보, Study Instance UID, Series Instance UID등이 포함되어 있다.
data = pydicom.read_file('/content/ID_000012eaf.dcm')Dataset.file_meta -------------------------------
(0002, 0000) File Meta Information Group Length UL: 188
(0002, 0001) File Meta Information Version OB: b'\x00\x01'
(0002, 0002) Media Storage SOP Class UID UI: CT Image Storage
(0002, 0003) Media Storage SOP Instance UID UI: 1.2.840.4267.32.337944818669776895705763408052798539612
(0002, 0010) Transfer Syntax UID UI: Explicit VR Little Endian
(0002, 0012) Implementation Class UID UI: 1.2.40.0.13.1.1.1
(0002, 0013) Implementation Version Name SH: 'dcm4che-1.4.35'
-------------------------------------------------
(0008, 0018) SOP Instance UID UI: ID_000012eaf
(0008, 0060) Modality CS: 'CT'
(0010, 0020) Patient ID LO: 'ID_f15c0eee'
(0020, 000d) Study Instance UID UI: ID_30ea2b02d4
(0020, 000e) Series Instance UID UI: ID_0ab5820b2a
(0020, 0010) Study ID SH: ''
(0020, 0032) Image Position (Patient) DS: [-125.000000, -115.897980, 77.970825]
(0020, 0037) Image Orientation (Patient) DS: [1.000000, 0.000000, 0.000000, 0.000000, 0.927184, -0.374607]
(0028, 0002) Samples per Pixel US: 1
(0028, 0004) Photometric Interpretation CS: 'MONOCHROME2'
(0028, 0010) Rows US: 512
(0028, 0011) Columns US: 512
(0028, 0030) Pixel Spacing DS: [0.488281, 0.488281]
(0028, 0100) Bits Allocated US: 16
(0028, 0101) Bits Stored US: 16
(0028, 0102) High Bit US: 15
(0028, 0103) Pixel Representation US: 1
(0028, 1050) Window Center DS: "30.0"
(0028, 1051) Window Width DS: "80.0"
(0028, 1052) Rescale Intercept DS: "-1024.0"
(0028, 1053) Rescale Slope DS: "1.0"
(7fe0, 0010) Pixel Data OW: Array of 524288 elements
- Dicom 파일 이해하기
CT image의 픽셀 값은 일반 RGB 이미지와는 다르다. RGB 이미지의 각 픽셀은 0 ~ 255의 값으로 이루어져 있지만, CT는 Hounsfield Unit (HU)라는 단위로 이루어져 있다. 여기서 HU란 X 선이 몸을 투과할 때 감쇠되는 정도를 나타내는 단위다.
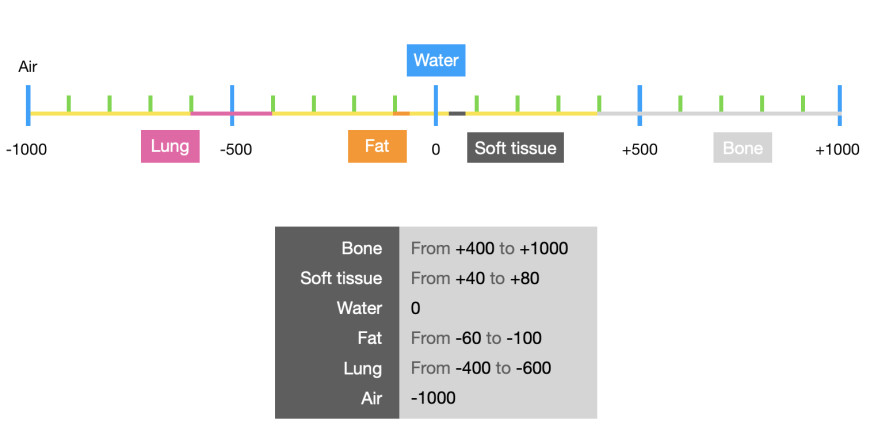
조금 더 이해하기 쉽게 표를 가지고 왔는데 , 다음 그림은 물 = 0을 기준으로 각 부위의 HU 값을 나타내는 표다.
CT 영상의 픽셀들은 HU 값으로 구성되어 있기 때문에 보고 싶은 신체 부위가 있다면 HU 표를 참고해 Window Center와 Window Width를 조절한 뒤에 그 부분 위주로 출력해줄 수 있다.
Window Center는 보고 싶은 부위의 HU 값을 의미하고, Window Width는 WC를 중심으로 관찰하고자 하는 HU 범위를 의미한다.
그래서 만약 뼈를 보고 싶다고 하면, window center 와 width을 400과 1000으로 넣고 스케일링 해주면 뼈 부분이 더 잘 보이게 된다.
- Dicom 파일의 window 조정 code
# dicom 파일에서 각 value값들 가져오기
def get_window(data):
dicom_fields = [data[('0028', '1050')].value, # Window Center
data[('0028', '1051')].value, # Window Width
data[('0028', '1052')].value, # Rescale Intercept
data[('0028', '1053')].value] # Rescale Slope
return [get_first_of_dicom_field_as_int(x) for x in dicom_fields]# 원하는 window값 넣어서 scaling 해주기
def window_image(img, window_center, window_width, intercept, slope, rescale=True):
img = (img * slope + intercept)
img_min = window_center - window_width//2
img_max = window_center + window_width//2
img[img<img_min] = img_min
img[img>img_max] = img_max
if rescale:
img = (img-img_min) / (img_max - img_min)
return img window_center, window_width, intercept, slope = get_window(data)
img = pydicom.read_file('/content/ID_000012eaf.dcm').pixel_array #array# displaying the image : Three window
# Brain window : 40-80
# Subdural window : 80~200
# Bone window : 600~2800
fig, (ax1, ax2, ax3, ax4) = plt.subplots(4,1, sharex='col', figsize=(10,24), gridspec_kw={'hspace': 0.1, 'wspace': 0})
ax1.set_title('Default window')
im1 = ax1.imshow(img, cmap=plt.cm.bone)
ax2.set_title('Brain window') # Brain window
img2 = window_image(img, 40, 80, intercept, slope)
im2 = ax2.imshow(img2, cmap=plt.cm.bone)
ax3.set_title('Subdural window')
img3 = window_image(img, 80, 200, intercept, slope) # Blood/Subdural window
im3 = ax3.imshow(img3, cmap=plt.cm.bone)
# ax3.annotate('', xy=(150, 380), xytext=(120, 430),
# arrowprops=dict(facecolor='red', shrink=0.05),
# )
# ax3.annotate('', xy=(220, 430), xytext=(190, 500),
# arrowprops=dict(facecolor='red', shrink=0.05),
# )
ax4.set_title('Bone window')
img4 = window_image(img, 600, 2800, intercept, slope) # Bone window
im4 = plt.imshow(img4, cmap=plt.cm.bone)
for ax in fig.axes:
ax.axis("off")
plt.show()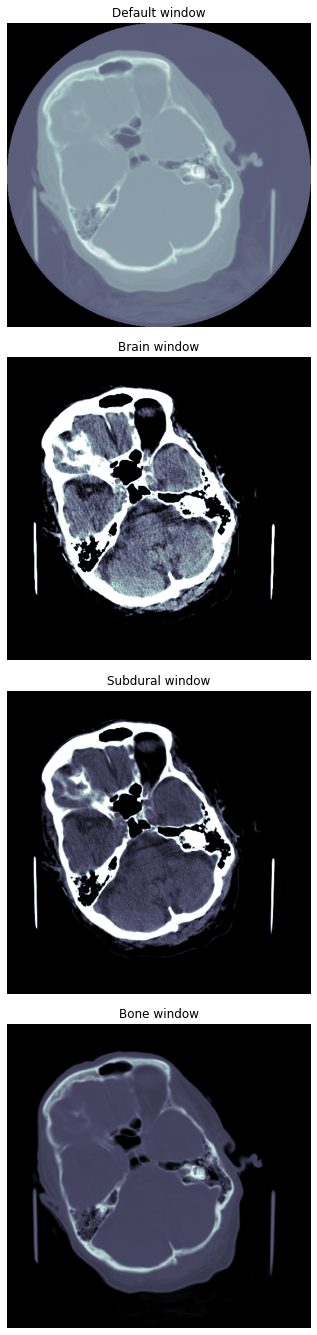
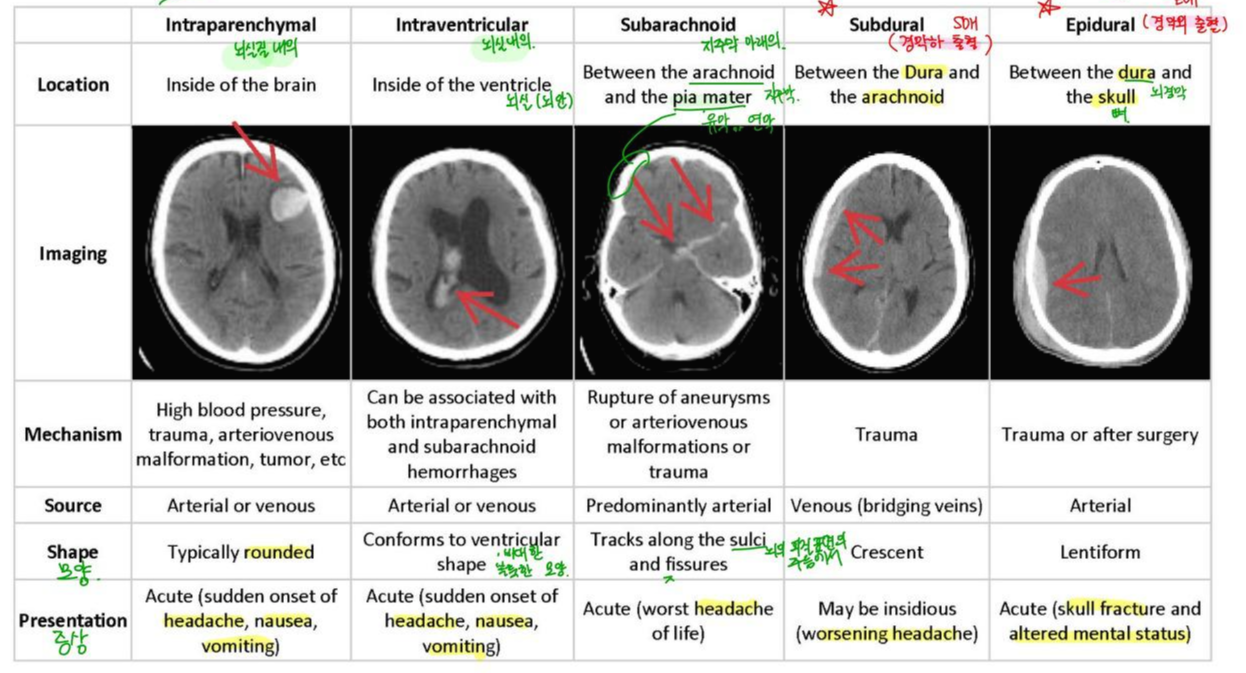
2. 뇌출혈 5가지 Subtypes
- cerebral parenchymal Hemorrhage(CPH) : 뇌 안에 생기는 출혈.
- intraventricular Hemorrhage(IVH) : 뇌실 안에 생기는 출혈.
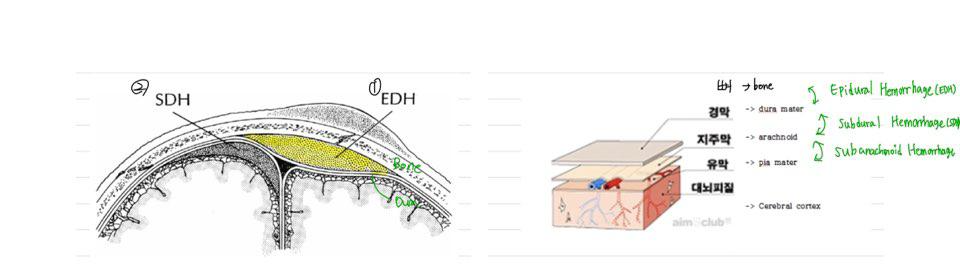
- subarachnoid Hemorrhage(SAH) : 지주막(arachnoid) 과 연막(pia mater) 사이에서 생기는 출혈
- subdural Hemorrhage(SDH) : 경막(dura)와 지주막(arachnoid) 사이에서 생기는 출혈.
- epidural Hemorrhage(EDH) : 경막(dura)와 뼈 사이에 생기는 출혈.
*뇌실 : 뇌 사이의 빈 공간.
[ 각 뇌출혈 종류의 특징들 ]
출처 : Kaggle - RNSA intracranial-hemorrhage-detection
아래 이미지는 공부하면서 필기해논 그림이다.
각 뇌출혈 위치, 이미지, 모양, 증상을 설명하고 있으니 참고 하실 분들은 참고하세요 :)
'Artificial Intelligence > Computer Vision' 카테고리의 다른 글
| Optical Flow 개념 및 알고리즘 종류 (0) | 2021.09.28 |
|---|---|
| pretrained model layer 수정하기 (1) | 2021.07.28 |

Action speaks louder than words. 하루 하루의 기록을 습관화 합니다 📖
포스팅이 좋았다면 "좋아요❤️" 또는 "구독👍🏻" 해주세요!
